
Jak komórki chronią swoje chromosomy — najnowsze odkrycia z Nature Communications
Wyniki badań zespołu naukowców Uniwersytetu Wrocławskiego, opublikowane w ostatnim czasie w Nature Communications, przybliżają nas do zrozumienia procesów leżących u podstaw transformacji nowotworowej komórek i mogą w przyszłości pomóc w lepszym wyjaśnieniu, w jaki sposób zaburzenia naprawy DNA przyczyniają się do rozwoju nowotworów u ludzi.
Komórki muszą wyjątkowo precyzyjnie kopiować i naprawiać DNA, aby nie doprowadzić do uszkodzeń materiału genetycznego. Szczególnie wrażliwym miejscem na chromosomach są centromery — regiony odpowiedzialne za prawidłowy rozdział chromatyd siostrzanych1 między komórkę matczyną a komórkę potomną podczas podziału komórki. Nawet niewielkie nieprawidłowości w tym obszarze mogą prowadzić do poważnych zaburzeń stabilności genomu.
W niedawno opublikowanej pracy w Nature Communications zespół Centrum Badawczego Naprawy DNA i Replikacji pod kierownictwem dr hab. Karola Kramarza pokazał, że specjalna modyfikacja białek naprawczych — tzw. poli-SUMOilacja — działa jak molekularny „hamulec”. Ogranicza ona rearanżacje materiału genetycznego w centromerach u drożdży Schizosaccharomyces pombe. Dzięki temu mechanizmowi komórka zapobiega powstawaniu niepożądanych zmian w strukturze chromosomów i chroni stabilność genomu. Warto podkreślić, że praca ta jest w całości efektem działalności Centrum Badawczego Naprawy DNA i Replikacji, a kluczową rolę w rozwoju projektu odegrała dr Katarzyna Markowska, pierwsza autorka publikacji.
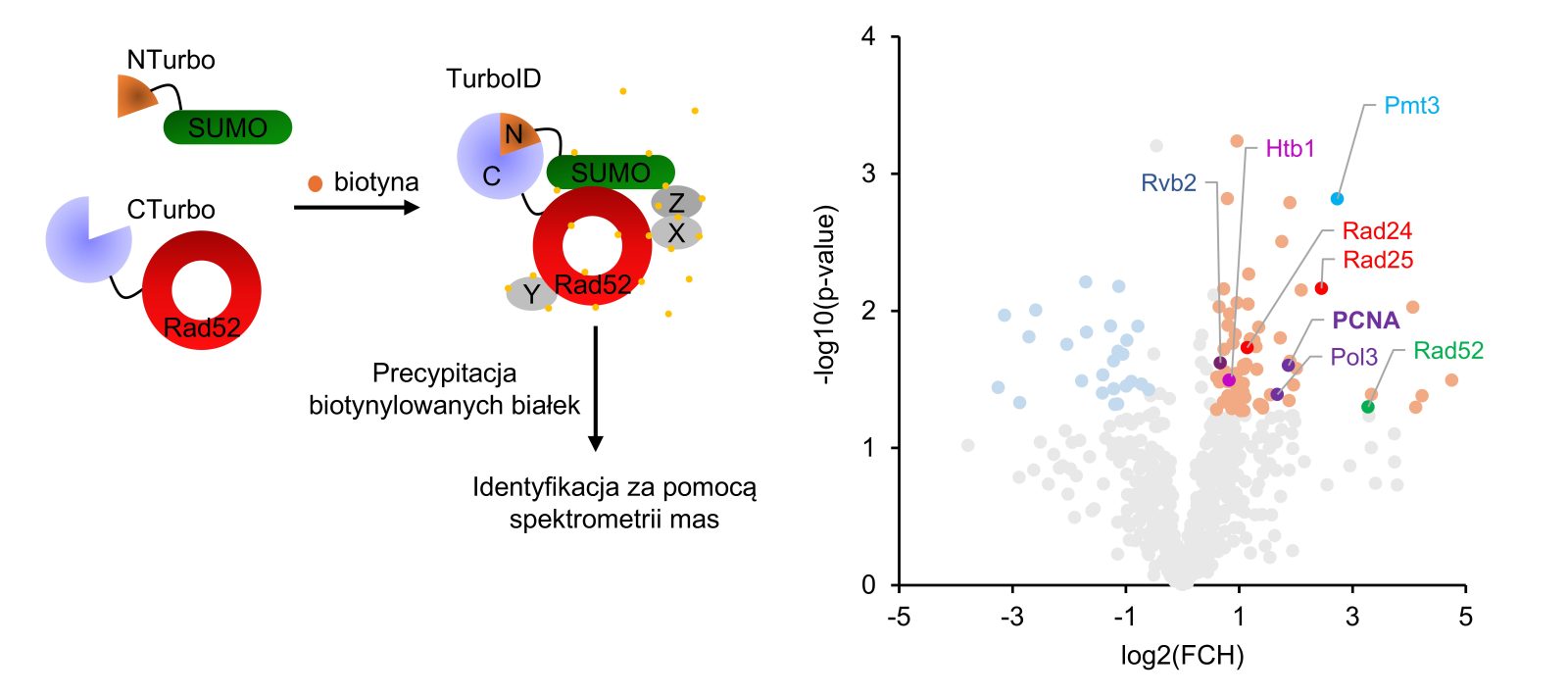
Zespół dr. hab. Karola Kramarza pokazał nowe funkcje białka SUMO (z ang. small ubiquitin-like modifier), małej cząsteczki podobnej do ubikwityny – białka obecnego u wszystkich organizmów eukariotycznych, w tym u ludzi, które reguluje działanie komórki, m.in. przez kierowanie innych białek na degradację lub zmianę ich właściwości i lokalizacji. SUMO jest kluczowym regulatorem podstawowych procesów związanych z metabolizmem DNA, zarówno u jednokomórkowych drożdży, jak i w komórkach ludzkich. SUMO, podobnie do ubikwityny, może formować polimeryczne łańcuchy, chociaż dotychczas wiązano tę modyfikację głównie ze szlakiem degradacji białek. Uzyskane przez zespół dane sugerowały rolę łańcuchów SUMO w organizacji struktury centromeru – złożonego z licznych powtórzeń fragmentu chromosomu, stanowiącego trudne do replikacji miejsce genomu. Dr hab. Ireneusz Litwin wprowadził do codziennego użytku technikę badania struktury DNA centromerów z wykorzystaniem sekwencjonowania następnej generacji, a dr Paulina Tomaszewska opracowała metodologię analiz bioinformatycznych uzyskanych danych. Dzięki temu możliwe było bezpośrednie pokazanie, że brak poliSUMOilacji prowadzi do zmian w organizacji centromeru. W pracy wykorzystano również nowoczesną technikę proteomiczną do badania lokalnej SUMOilacji, której wprowadzenie umożliwiła praca ówczesnej magistrantki w Zespole, Julii Kończak. Analizy składu SUMOilowanych białek w miejscach naprawy DNA wykonał dr Michał Tracz z Wydziału Biotechnologii, dzięki pozyskanemu w ramach programu IDUB spektrometrowi masowemu. Doprowadziło to do identyfikacji białka PCNA (z ang. proliferating cell nuclear antygen), kluczowego czynnika replikacyjnego. Analizę SUMOilacji PCNA wykonała doktorantka Dorota Misiorna, wskazując jednocześnie, że poliSUMOilacja tego białka wpływa na aktywację odpowiednich ścieżek naprawy, które komórka wykorzystuje do wznawiania zatrzymanego procesu syntezy DNA. Brak łańcuchów SUMO na PCNA prowadzi do zwiększonej rekombinacji w centromerach, co może zaburzać ich strukturę. Bezpośrednie pomiary poziomów rekombinacji w centromerach przeprowadziła doktorantka Aleksandra Bogdańska. Wreszcie, badania wykonane przez studenta realizującego program studiów magisterskich w ramach wymiany Erasmus+, Miki Haenen, dotyczące wpływu sztucznego łańcucha SUMO na jednym z białek naprawczych, przyczyniły się do lepszego zrozumienia mechanizmu rekombinacji w centromerze.
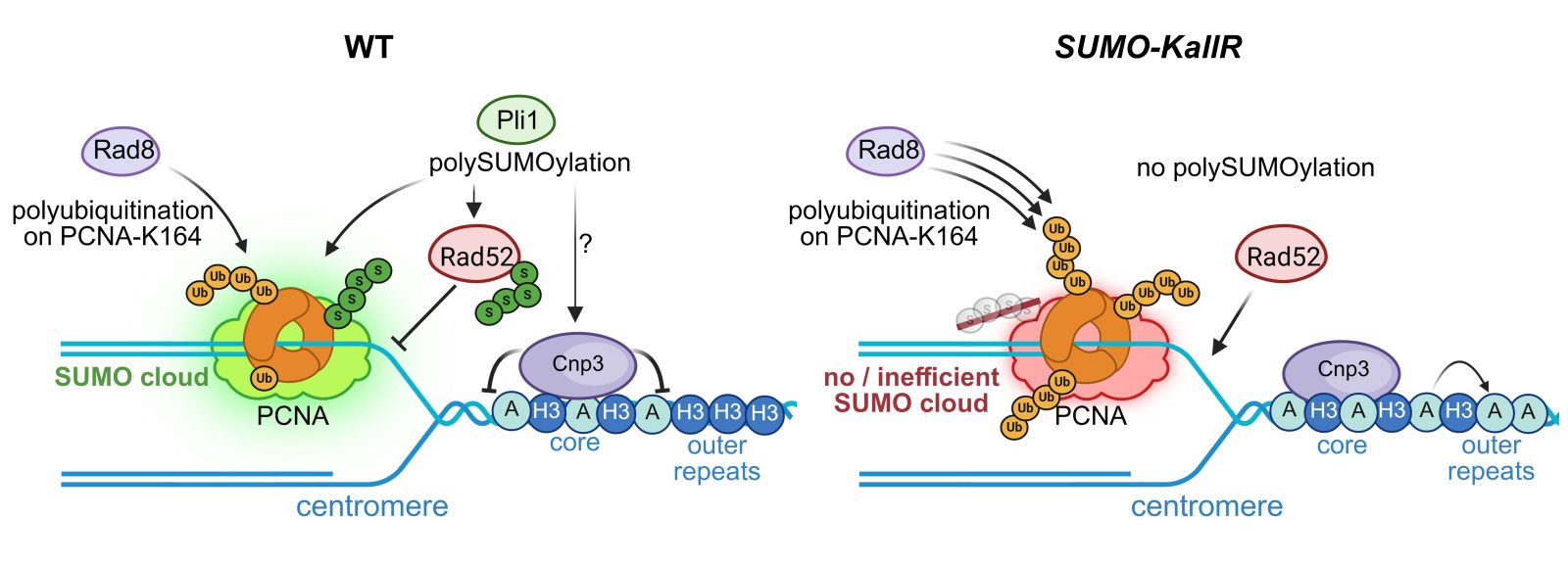
W efekcie, zespół dr. hab. Karola Kramarza dowiódł, że poliSUMOilacja, wcześniej kojarzona głównie z kierowaniem modyfikowanych białek na ścieżkę degradacji, pełni istotną rolę w ochronie centromerów przed nadmierną rekombinacją. Mówiąc bardziej szczegółowo, badacze pokazali, że poli-SUMOilacja kluczowego białka biorącego udział w replikacji DNA, PCNA, działa jak „bezpiecznik”, który zapobiega nadmiernemu dołączaniu ubikwityny do tego białka. Od tego zależy, w jaki sposób komórka wznawia zatrzymaną syntezę DNA w obszarze centromerów. Jeśli poli-SUMOilacji brakuje, dochodzi do niewłaściwej aktywacji ścieżek naprawy DNA, co zwiększa rekombinację w centromerach i może zaburzać ich prawidłową strukturę.
Łańcuchy SUMO pomagają utrzymać stabilność genomu w warunkach normalnych, w których występuje stały, niewielki poziom stresu, czyli w sytuacji, w jakiej znajdują się codziennie ludzkie komórki. Niekontrolowana rekombinacja w normalnych warunkach prowadzi do rearanżacji genomu, a to może leżeć u podstaw transformacji nowotworowej komórek ludzkich. Centromery na chromosomach ludzkich stanowią wrażliwe miejsca w genomie, ulegające częstym rearanżacjom. Otrzymane wyniki przybliżają nas zatem do zrozumienia procesów leżących u podstaw transformacji nowotworowej komórek i mogą w przyszłości pomóc w lepszym wyjaśnieniu, w jaki sposób zaburzenia naprawy DNA przyczyniają się do rozwoju nowotworów u ludzi.
Link do publikacji PolySUMOylation of PCNA and Rad52 restricts centromeric recombination in fission yeast.
***
1chromatydy siostrzane – identyczne kopie tej samej cząsteczki DNA, powstałe w wyniku replikacji chromosomu, połączone ze sobą w obszarze centromeru.
Data publikacji: 10.12.2025 r.
Dodane przez: E.K.



